Ferramentas pedométricas
Ferramentas pedométricas para análise de pedologia quantitativa
 Image credit: Elias
Image credit: Elias
Introdução
Nesse tutorial serão abordados através do Algoritmo para Pedologia Quantitativa (AQP em inglês) e pacotes auxiliares, uma coleção de códigos, ideias, e plotes usando o software R (R Core Team, 2019). Toda a teoria por trás de grande parte do código pode ser encontrada no artigo Algorithms for quantitative pedology: A toolkit for soil scientists (Beaudette et al., 2013). Além disso alguns exemplos com aplicações das ferramentas podem ser encontrados em Pinheiro et al. (2016) e Pinheiro et al. (2018). Para fins didáticos o tutorial foi dividido em três partes e os pacotes usados serão soilDB, que contém uma base de dados de solos para análises pedométricas, AQP que contém as principais ferramentas de pedologia quantitativa usadas no tutorial, sharpshootR que contém uma coelção de funções que dão suporte ao levantamento e exploração de dados de solo, SP um pacote que fornece classes e métodos para dados espaciais: pontos, linhas, polígonos e grades, Hmisc que contém muitas funções úteis para análise de dados, gráficos de alto nível, operações utilitárias, funções para calcular o tamanho de amostras, importar e manipular conjuntos de dados, imputar valores ausentes, criação avançada de tabelas, análise de cluster entre outras funções, lattice que é um sistema de visualização de dados de alto nível, poderoso e elegante, com ênfase em dados multivariados, MASS com funções e conjuntos de dados para apoiar Venables e Ripley, Modern Applied Statistics with S, plyr que contém um conjunto de ferramentas que resolve problemas tais como: Divisão, aplicação e combinação de dados e cluster que é um pacote com métodos para análise de componentes principais (PCA) e cluster a fim de encontrar grupos em conjunto de dados.
Base de dados dos exercícios
Todos os dados usados nos exercícios ou são do pacote AQP ou são do pacote soilDB. Muitas das informações contidas nesse tutorial foram extraídas do projeto do AQP e todo crédito é dado ao autor.
# Carregando os pacotes no R
library(soilDB)
library(aqp)
library(sharpshootR)
library(sp)
library(Hmisc)
library(lattice)
library(MASS)
library(plyr)
library(cluster)Parte I
Na parte I serão abordadas características das funções básicas do AQP, conceito de coleção de perfis de solos, SoilProfileCollection, e verificação da estrutura de dados na coleção, além de alguns plotes básicos. A classe SoilProfileCollection foi projetada para simplificar o processo de trabalhar com a coleção de dados associados aos perfis de solo: dados em nível de local (site), dados em nível de horizonte, dados espaciais, dados de horizontes de diagnósticos, metadados e etc. Os exemplos listados abaixo devem ser copiados/colados deste documento e executados de forma interativa no R. Lembrando que antes de executar você deve instalar e carregar todas as bibliotecas (pacotes) mencionados.
Criação de Objetos
Os objetos SoilProfileCollection geralmente são criados através de objetos data.frame (tabelas retangulares de dados) que contêm pelo menos três colunas essenciais:
- Uma coluna de identificação que identifica exclusivamente grupos de horizontes, um perfil de solo (por exemplo, pedons)
- Limites superiores do horizonte
- Limites inferiores do horizonte
O data.frame deve ser pré-classificado de acordo com o ID do perfil e o limite superior do horizonte. A notação de fórmula é usada para definir as colunas usadas para promover um objeto data.frame.
# Carregando dados de solos
# upgrade para SoilProfileCollection
# 'id' é o nome da coluna que contém o ID do perfil
# 'top' é o nome da coluna que contém os limite superior do horizonte
# 'bottom' é o nome da coluna que contém os limite inferior do horizonte
data(sp1) # Conjunto de dados sp4 do pacote AQP (data.frame)
aqp::depths(sp1) <- id ~ top + bottom
aqp::site(sp1) <- ~ group Acessando, configurando e substituindo dados
As funções de acessar são usadas para extrair componentes específicos dos objetos SoilProfileCollection.
# Métodos para inspeção dos objetos
aqp::idname (sp1) # auto-explicativo## [1] "id"aqp::horizonDepths(sp1) # auto-explicativo## [1] "top" "bottom"aqp::depth_units(sp1) # defaults 'cm'## [1] "cm"aqp::metadata(sp1) # ## $aqp_df_class
## [1] "data.frame"
##
## $aqp_group_by
## [1] ""
##
## $aqp_hzdesgn
## [1] ""
##
## $aqp_hztexcl
## [1] ""
##
## $depth_units
## [1] "cm"
##
## $stringsAsFactors
## [1] FALSE
##
## $original.order
## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25
## [26] 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50
## [51] 51 52 53 54 55 56 57 58 59 60aqp::profile_id(sp1) # vector com IDs dos perfis## [1] "P001" "P002" "P003" "P004" "P005" "P006" "P007" "P008" "P009"aqp::horizonNames(sp1) # coluna com osnomes dos dados dos horizontes## [1] "id" "top" "bottom" "bound_distinct"
## [5] "bound_topography" "name" "texture" "prop"
## [9] "structure_grade" "structure_size" "structure_type" "stickiness"
## [13] "plasticity" "field_ph" "hue" "value"
## [17] "chroma" "hzID"aqp::siteNames(sp1) # coluna com os nomes do dados dos locais (site data)## [1] "id" "group"length(sp1) # número de perfis na coleção## [1] 9nrow(sp1) # número de horizontes na coleção## [1] 60names(sp1) # nomes de colunas dos dados do site e do horizonte, concatenados em um único vetor## horizons1 horizons2 horizons3 horizons4
## "id" "top" "bottom" "bound_distinct"
## horizons5 horizons6 horizons7 horizons8
## "bound_topography" "name" "texture" "prop"
## horizons9 horizons10 horizons11 horizons12
## "structure_grade" "structure_size" "structure_type" "stickiness"
## horizons13 horizons14 horizons15 horizons16
## "plasticity" "field_ph" "hue" "value"
## horizons17 horizons18 site
## "chroma" "hzID" "group"min(sp1) # profundidade de perfil mais raso da coleção## [1] 59max(sp1) # profundidade de perfil mais profundo da coleção## [1] 240Dados do horizonte e do site (local)
Normalmente, os dados do horizonte e do site são os componentes mais importantes dos objetos SoilProfileCollection. Ambos são armazenados internamente como objetos data.frame; com uma ou mais linhas (por ID do perfil) na tabela do horizonte e uma linha (por ID do perfil) na tabela do site. As colunas de qualquer tabela podem ser acessadas com a notação $. Novos dados podem ser atribuídos a qualquer tabela da mesma maneira, desde que o comprimento dos novos dados sejam:
- Mesmo tamanho que o número de perfis na coleção (o destino é a tabela de sites).
- Mesmo comprimento que o número de horizontes na coleção (o destino é a tabela do horizonte).
Plotando SoilProfileCollection
O método plot() para objetos SoilProfileCollection gera esboços de perfis na coleção com base nos limites dos horizontes, alinhados verticalmente a uma sequência inteira de um ao número total de perfis. Os nomes dos horizontes são extraídos automaticamente de um nome de atributo no nível do horizonte (se presente) ou por meio de um atributo alternativo fornecido como argumento: name = 'nomedacoula'. As cores dos horizontes são geradas automaticamente a partir do atributo no nível do horizonte soil_color, ou qualquer outro atributo da descrição de cor compatível com R, fornecido como argumento: color = 'nomedacoula'. Essa função é altamente personalizável; portanto, é prudente consultar a ajuda (plotSPC) de tempos em tempos. As cores do solo na notação Munsell podem ser convertidas em cores compatíveis com R via munsell2rgb().
data(sp4) # Conjunto de dados sp4 do pacote AQP (data.frame)
aqp::depths(sp4) <- id ~ top + bottom
# Atribuição de novos dados a atributos existentes ou novos
sp4$elevation <- rnorm(n=length(sp4), mean=1000, sd=150) # no nível do site, com base no comprimento dos dados atribuídos
sp4$thickness <- sp4$bottom - sp4$top # nível do horizonte
# Extração de atributos específicos por nome
sp4$clay # Vetor de conteúdo de argila (dados do horizonte)## [1] 21 27 32 55 25 34 3 15 27 32 25 31 33 13 21 23 15 17 12 19 14 14 22 25 40
## [26] 51 67 24 25 32Os dados do horizonte e do local (site) também podem ser modificados por extração para um data.frame, seguidos pela substituição (dados do horizonte) ou junção (dados do site). Observe que, embora essa abordagem ofereça a maior flexibilidade, ela também é a mais perigosa. A substituição dos dados do horizonte por novos dados que não estão exatamente de acordo com a classificação original pode corromper seu SoilProfileCollection.
# extrair dados os horizontes para o data.frame
h <- aqp::horizons(sp4)
# Adicionar uma nova coluna e salvar de volta no objeto
h$random.numbers <- rnorm(n=nrow(h), mean=0, sd=1)
# repor dados originais dos horizontes com versão modificada
#!a ordem das linhas não deve ser alterada!
horizons(sp4) <- h
# extrair dados do site para o data.frame
s <- aqp::site(sp4)
# Adicionar um grupo para o site fake
s$group <- base::factor(rep(c('A', 'B'), length.out=nrow(s)))
# junte novos dados do site com dados anteriores: os dados antigos não são substituídos
site(sp4) <- s
# Verificar
sp4## SoilProfileCollection with 10 profiles and 30 horizons
## profile ID: id | horizon ID: hzID
## Depth range: 16 - 49 cm
##
## ----- Horizons (6 / 30 rows | 10 / 16 columns) -----
## id hzID top bottom name K Mg Ca CEC_7 ex_Ca_to_Mg
## colusa 1 0 3 A 0.3 25.7 9.0 23.0 0.35
## colusa 2 3 8 ABt 0.2 23.7 5.6 21.4 0.23
## colusa 3 8 30 Bt1 0.1 23.2 1.9 23.7 0.08
## colusa 4 30 42 Bt2 0.1 44.3 0.3 43.0 0.01
## glenn 5 0 9 A 0.2 21.9 4.4 18.8 0.20
## glenn 6 9 34 Bt 0.3 18.9 4.5 27.5 0.20
## [... more horizons ...]
##
## ----- Sites (6 / 10 rows | 3 / 3 columns) -----
## id elevation group
## colusa 1005.8124 A
## glenn 930.8280 B
## kings 1185.8329 A
## mariposa 793.5652 B
## mendocino 1148.8530 A
## napa 1176.2684 B
## [... more sites ...]
##
## Spatial Data:
## [EMPTY]# plot com separação de grupos
aqp::groupedProfilePlot(sp4, groups='group', max.depth=60, group.name.offset = -5, id.style='side')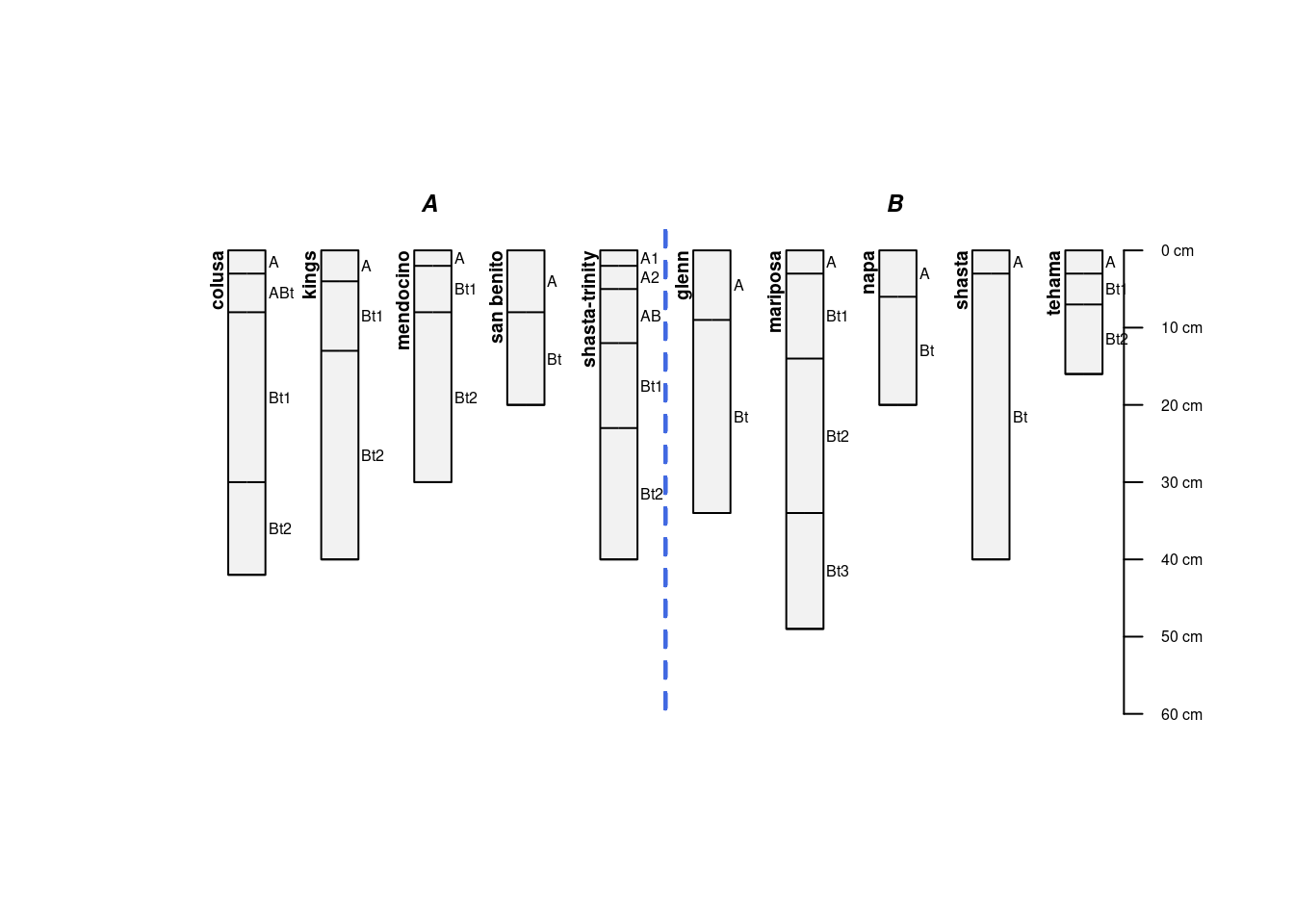
# Esboços Temáticos
sp4$clay # ## [1] 21 27 32 55 25 34 3 15 27 32 25 31 33 13 21 23 15 17 12 19 14 14 22 25 40
## [26] 51 67 24 25 32par(mar=c(1,1,3,1)) # tighter figure margins
plot(sp4, name='name', color='clay',cex.names=0.5,cex.id=0.5, id.style='top',col.legend.cex=1,family="serif", col.label='Argila', )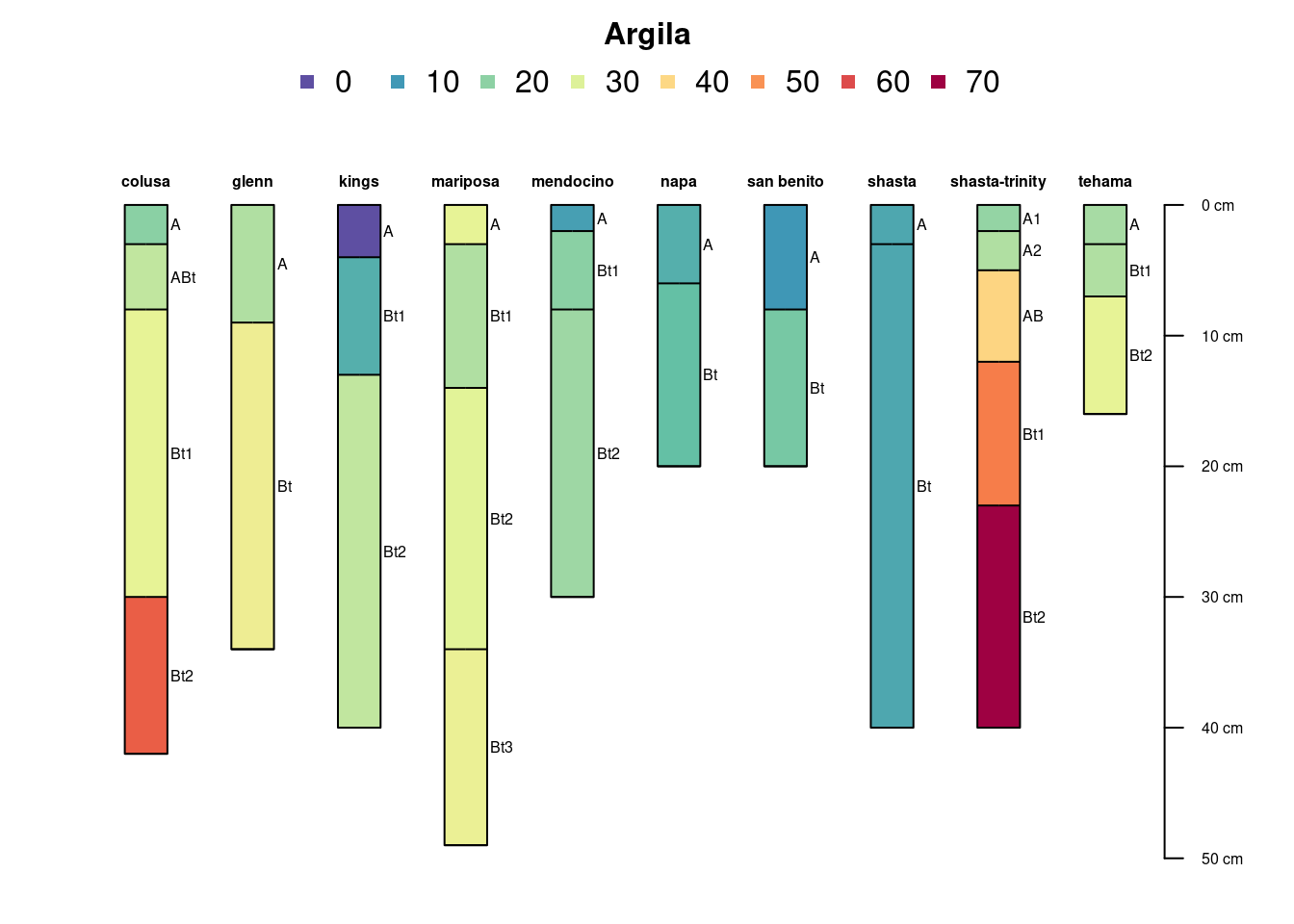
sp4$Ca # ## [1] 9.0 5.6 1.9 0.3 4.4 4.5 1.4 7.0 4.4 5.8 6.2 6.2 4.4 2.2 3.4
## [16] 3.7 24.1 21.5 0.7 5.6 3.5 2.0 6.6 4.1 3.5 1.5 0.8 16.3 16.5 13.7par(mar=c(1,1,3,1)) # tighter figure margins
plot(sp4, name='name', color='Ca',cex.names=0.5,cex.id=0.5, id.style='top',col.legend.cex=1,
col.label='Ca', family="serif")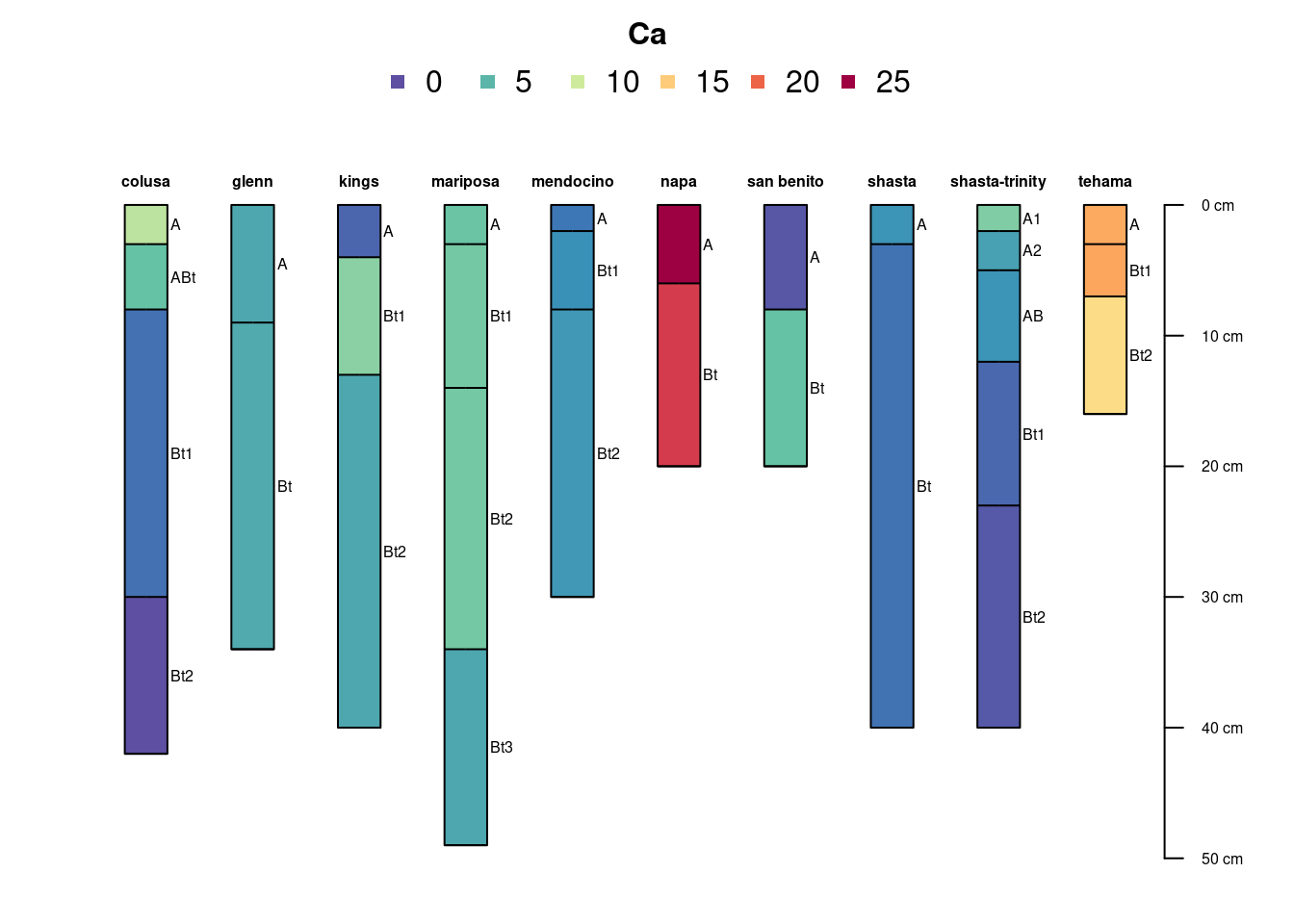
sp4$name # ## [1] "A" "ABt" "Bt1" "Bt2" "A" "Bt" "A" "Bt1" "Bt2" "A" "Bt1" "Bt2"
## [13] "Bt3" "A" "Bt1" "Bt2" "A" "Bt" "A" "Bt" "A" "Bt" "A1" "A2"
## [25] "AB" "Bt1" "Bt2" "A" "Bt1" "Bt2"par(mar=c(2,2,3,2)) # tighter figure margins
plot(sp4, name='name', color='name', col.palette=RColorBrewer::brewer.pal(9, 'Set1'), col.label='Nomes dos Horizontes',
cex.names=0.5)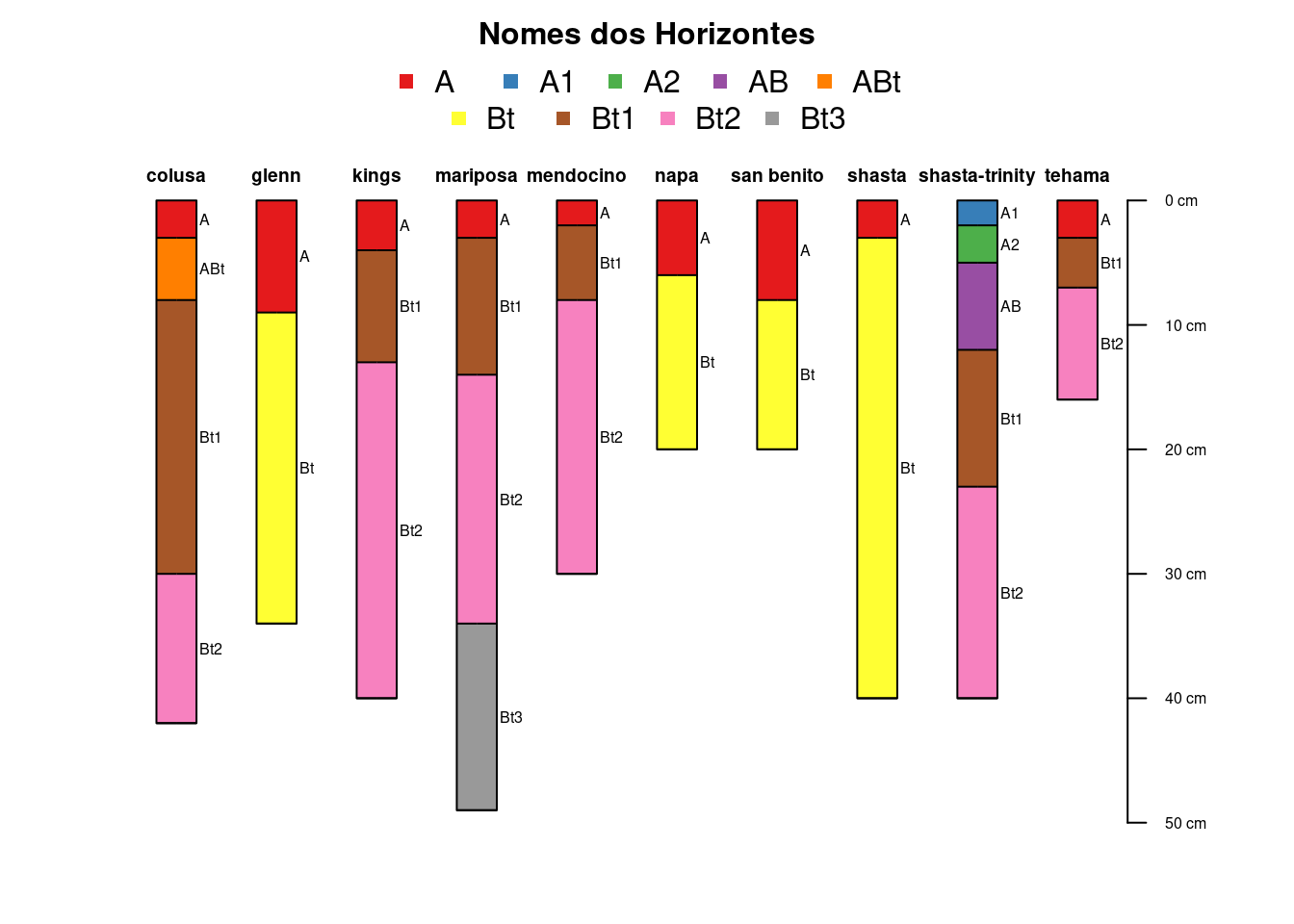
###############
### Boxplot ###
# Aqui usaremos o conjunto sp1
sp1$name1 <- aqp::generalize.hz(sp1$name,
new=c('A','AB', 'BA','B','BC', 'C', 'E', 'AE', 'O', 'H', 'R', '2C', '3C', '3B', '3A'),
pat=c('^A','^AB','^BA','^B','^BC','^C', '^E', '^AE', '^O', '^H', '^R', '^2C', '^3C','^3B', '^3A' ))
# compute horizon mid-points
sp1$mid <- with(horizons(sp1), (top+ bottom)/2)
# sort horizon designation by group-wise median values
hz.designation.by.median.depths <- names(sort(tapply(sp1$mid, sp1$name1, median)))
# plot the distribution of horizon mid-points by designation
lattice::bwplot(mid ~ factor(name, levels=hz.designation.by.median.depths),
data=horizons(sp1),
ylim=c(200, 0), ylab='Profundidade do ponto médio do horizonte (cm)',
scales=list(y=list(tick.number=10)),
panel= function ( ... ) {
panel.abline(h=seq(0, 180, by=10), v=1:length(hz.designation.by.median.depths), col=grey(0.8), lty=3)
panel.bwplot( ... ) })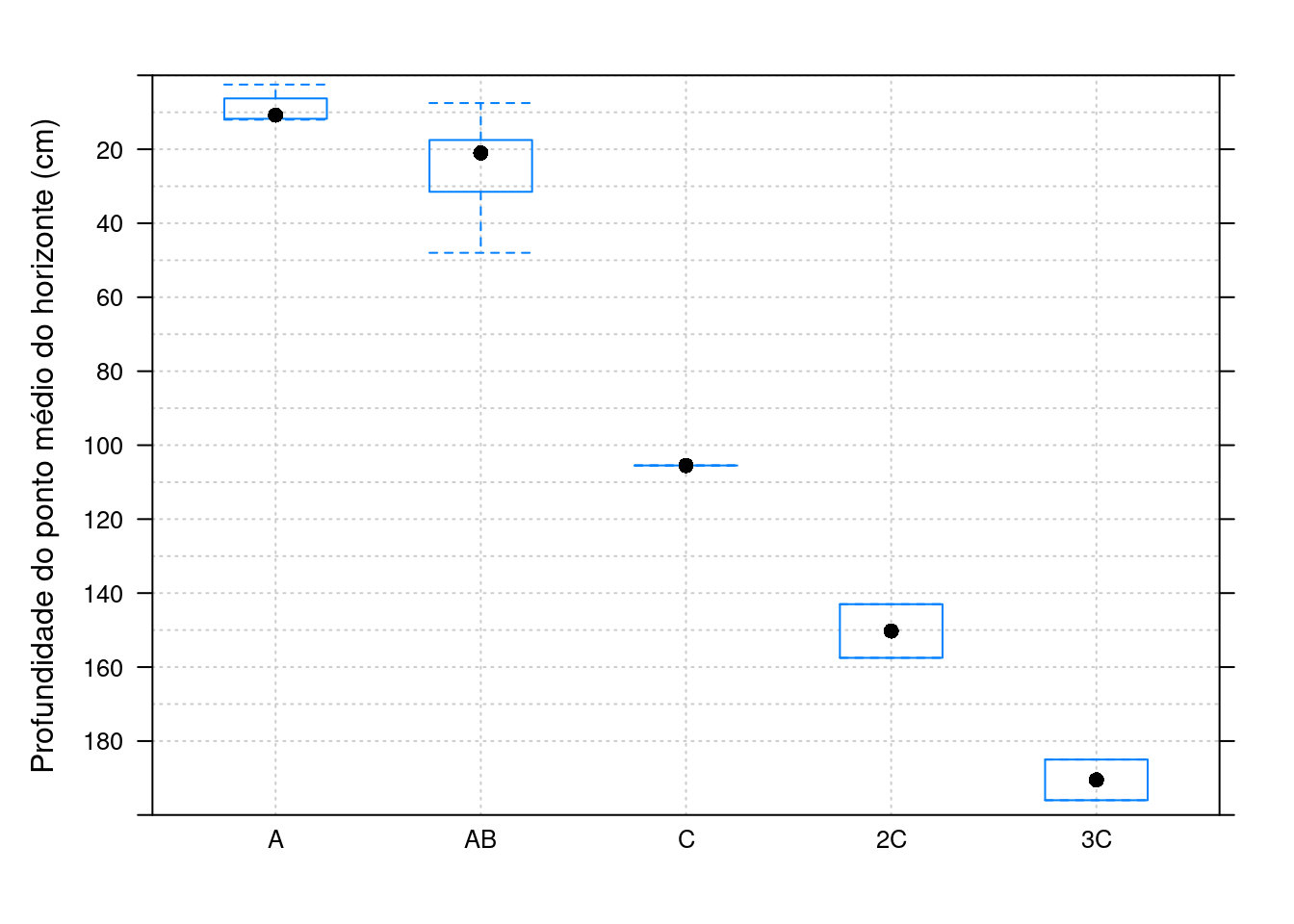
lattice::bwplot(prop~ factor(name1, levels=hz.designation.by.median.depths),
data=horizons(sp1),
ylab='Profundidade',
scales=list(y=list(tick.number=10)),
panel= function ( ... ) {
panel.abline(h=seq(0, 600, by=50), v=1:length(hz.designation.by.median.depths), col=grey(0.8), lty=3)
panel.bwplot( ... ) })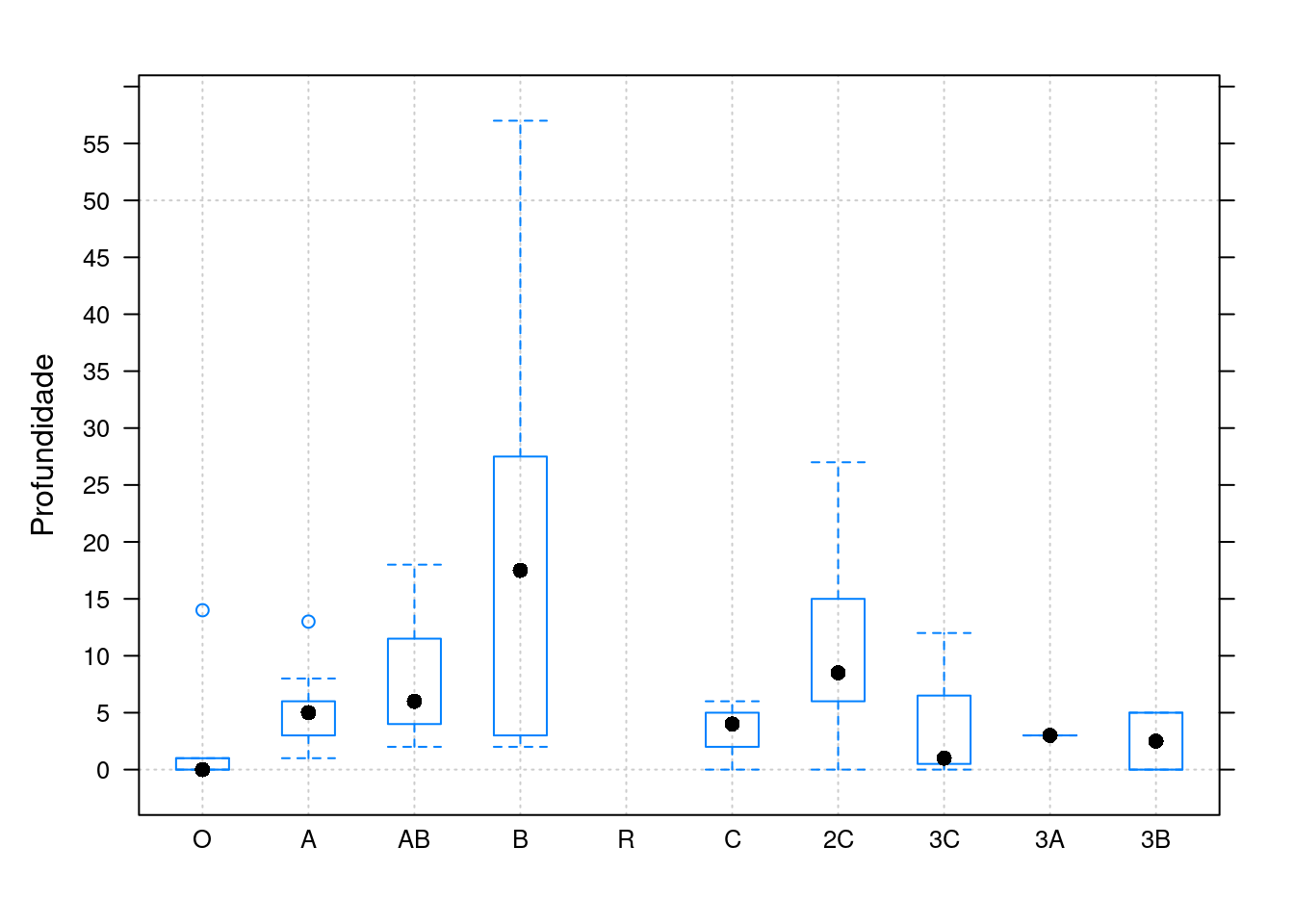
Parte II
Funções de fatiamento (slice)
Coleções de perfis de solo podem ser fatiadas (ou reamostradas) em intervalos regulares de profundidade com a função slice(). A estrutura de fatiamento e as variáveis de interesse são definidas por meio de notação de fórmula: seq ~ var.1 + var.2 + var.3 + … onde seq é uma sequência de números inteiros [por exemplo, 0:15 ou c (5,10,15,20) etc.] e var.1 + var.2 + var.3 + … são atributos no horizonte que se deseja fatiar. As variáveis contínuas e categóricas podem ser nomeadas no lado direito da fórmula. Os resultados retornados pela função slice () são SoilProfileCollection ou data.frame quando chamados com o argumento opcional just.the.data = TRUE. Por exemplo, para dividir nosso conjunto de dados de amostra em intervalos de 1 cm, de 0 a 15 cm:
# fatiar dados
sliced <- aqp::slice(sp4, fm= 0:50 ~ sand + silt + clay)
# verificar o resultado
class(sliced)## [1] "SoilProfileCollection"
## attr(,"package")
## [1] "aqp"# fatiar de 0 até a profundidade máxima dos perfis da coleção
sliced <- aqp::slice(sp4, fm= 0:max(sp4) ~ sand + silt + clay)
# Extraia todos os dados na faixa de 5--10 cm:
plot(sliced[, 5:10])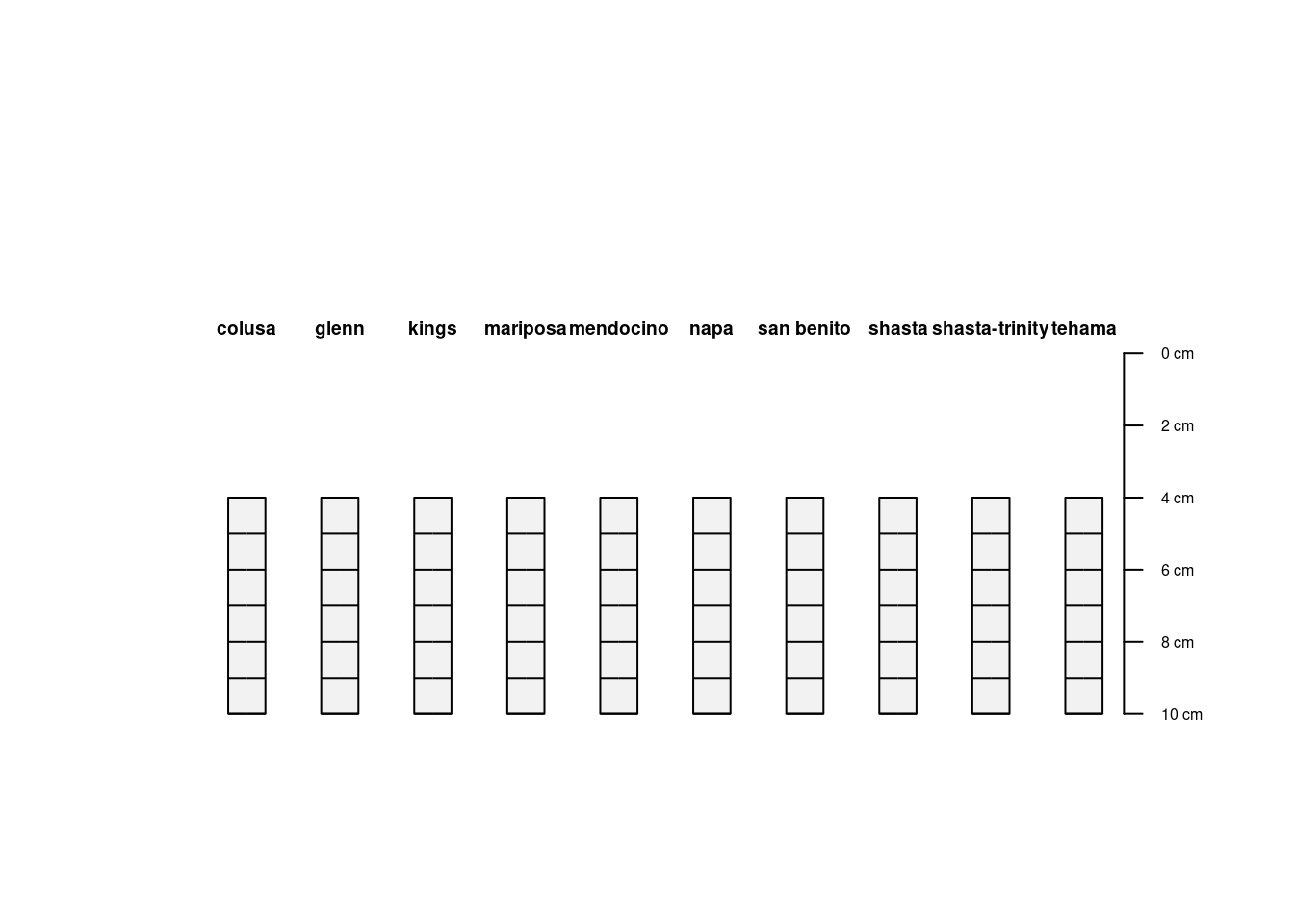
# Extraia todos os dados na faixa de 25 a 50 cm:
plot(sliced[, 25:50])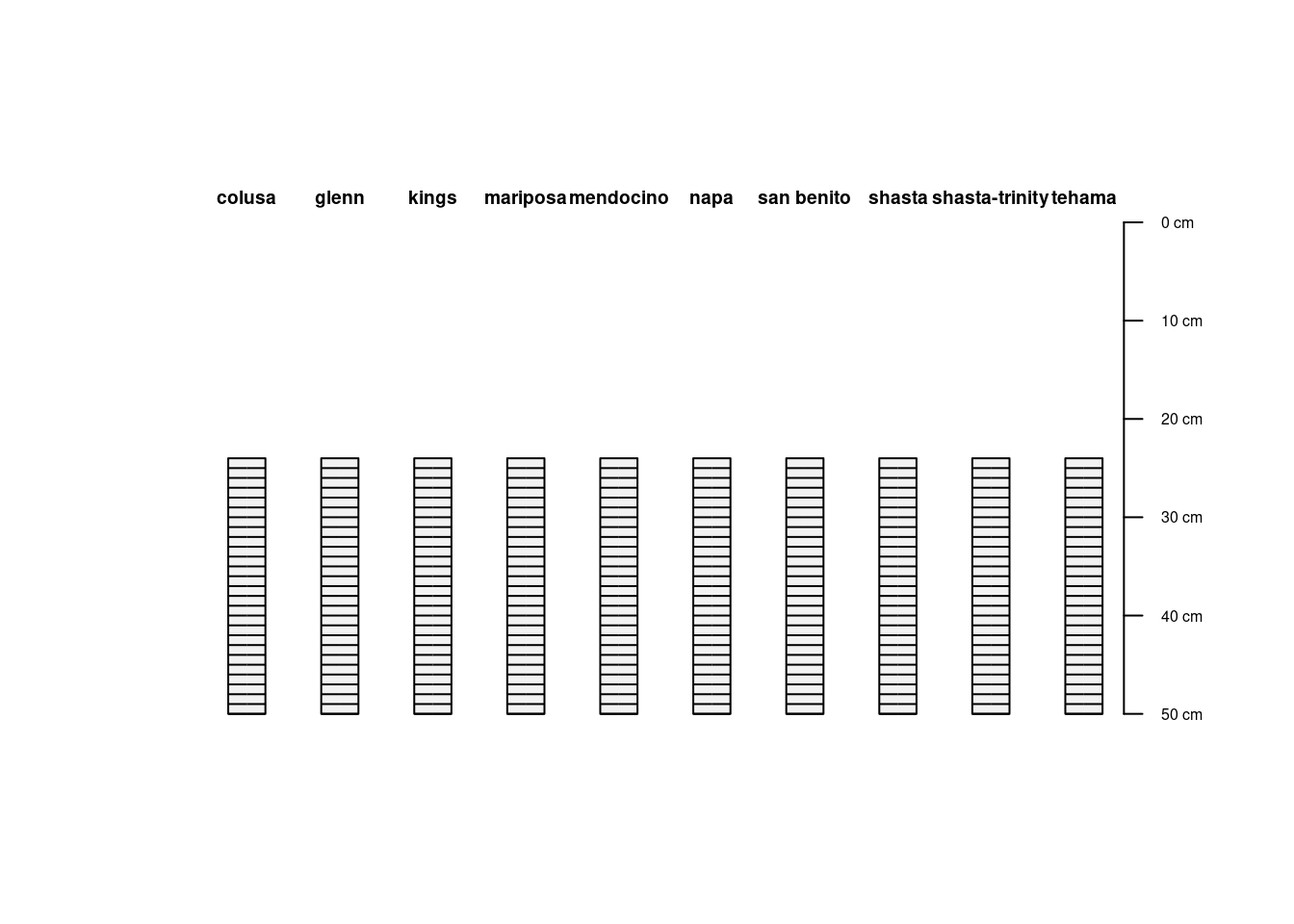
# Extraia todos os dados na faixa de 0--5, 10--20 e 40--50 cm:
plot(sliced[, c(0:5, 10:20, 40:50)])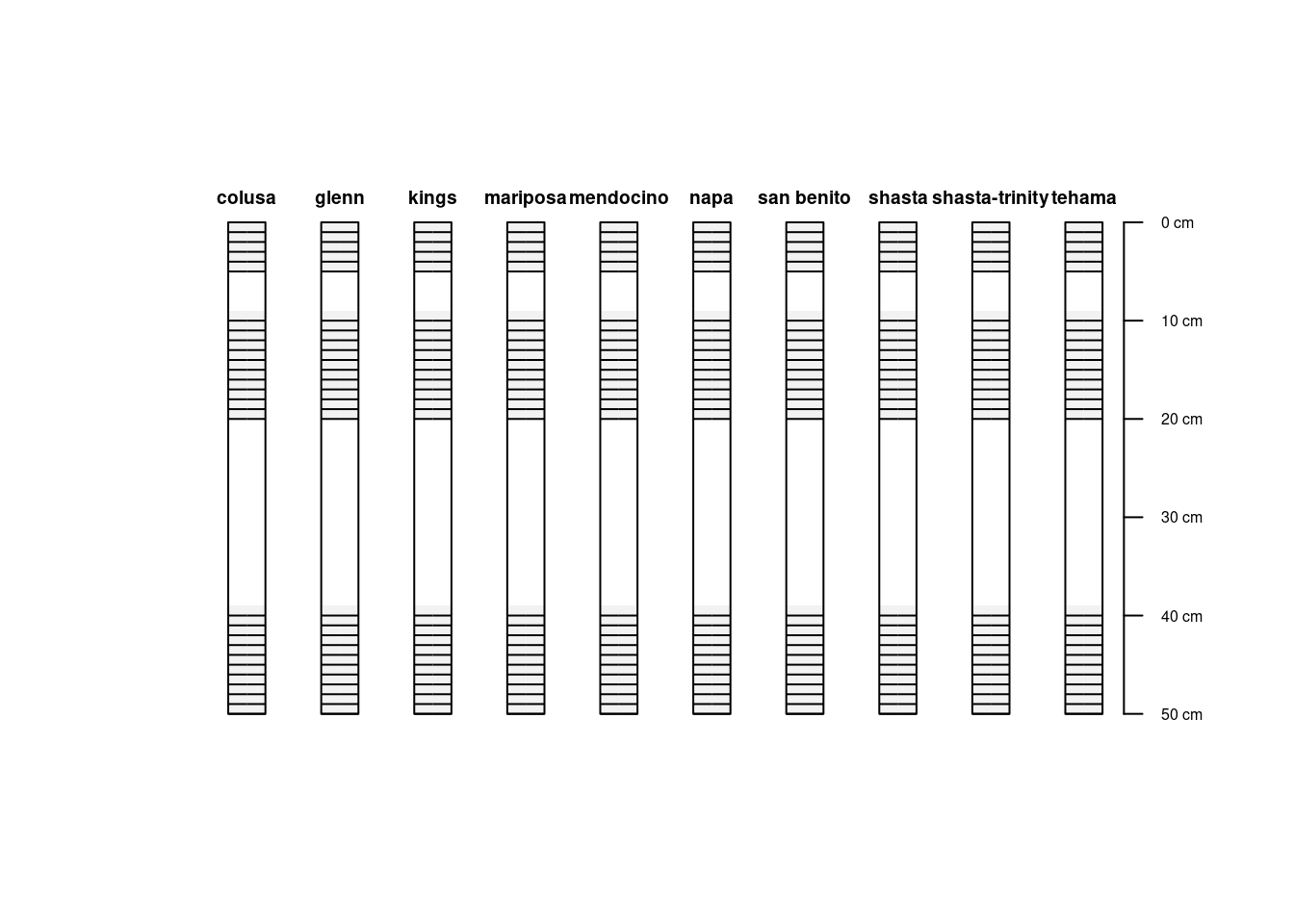
Agregando coleções de perfis de solo em “slabs” regulares
O resumo detalhado dos atributos no nível de horizonte é realizado com a função slab(). Os critérios de agrupamento de perfis e a seleção de atributos do horizonte são parametrizados por meio da fórmula: grupos ~ var1 + var2 + var3 onde variáveis nomeadas são agregadas em grupos ou onde variáveis nomeadas são agregadas em toda a coleção com fórmula: coleção ~ var1 + var2 + var3. A função de resumo padrão (slab.fun) calcula os percentis 5, 25, 50, 75 e 95 através do estimador de quantil Harrell-Davis.
A estrutura de profundidade (“slabs”) sobre a qual os resumos são calculados é definida com o argumento slab.structure usando:
- Um único número inteiro (por exemplo, 10): os dados são agregados em uma sequência regular de slabs de 10 unidades de espessura.
- Um vetor de 2 números inteiros [por exemplo, c (50, 60)]: os dados são agregados em profundidades que variam de 50 a 60 unidades.
- Um vetor de 3 ou mais números inteiros [por exemplo, c (0, 5, 10, 50, 100)]: os dados são agregados nas profundidades que variam de 0 a 5, 5 a 10, 10 a 50, 50 a 100 unidades. Essa é por exemplo umas das formas usadas para harmonização de dados em profundidades pré-definidas como é o caso do projeto GlobalSoilMap project (Arrouays et al., 2017) que é de 0-5, 5-15, 15-30, 30-60, 60-100, 100-200 cm.
# Agregar coleções de perfis de solo em “slabs” (conjuntos) regulares
# calculando o valor médio ignorando os dados ausentes
aqp::slab(sp4, fm= ~ sand + silt + clay, slab.structure=c(0,10), slab.fun=mean, na.rm=TRUE)## variable all.profiles value top bottom contributing_fraction
## 1 sand 1 47.63 0 10 1
## 2 silt 1 31.15 0 10 1
## 3 clay 1 21.11 0 10 1# Novamente, desta vez em grupos definidos por um atributo no nível do site:
aqp::slab(sp4, fm= group ~ sand + silt + clay, slab.structure=c(0,10), slab.fun=mean, na.rm=TRUE)## variable group value top bottom contributing_fraction
## 1 sand A 48.26 0 10 1
## 2 silt A 31.52 0 10 1
## 3 clay A 20.30 0 10 1
## 4 sand B 47.00 0 10 1
## 5 silt B 30.78 0 10 1
## 6 clay B 21.92 0 10 1# Novamente, desta vez em várias faixas de profundidade
aqp::slab(sp4, fm= ~ sand + silt + clay, slab.structure=c(0,5,15,30,60), slab.fun=mean, na.rm=TRUE)## variable all.profiles value top bottom contributing_fraction
## 1 sand 1 48.54000 0 5 1.0000000
## 2 sand 1 45.96000 5 15 1.0000000
## 3 sand 1 39.29310 15 30 0.7733333
## 4 sand 1 30.36923 30 60 0.3421053
## 5 silt 1 32.40000 0 5 1.0000000
## 6 silt 1 29.15000 5 15 1.0000000
## 7 silt 1 30.37069 15 30 0.7733333
## 8 silt 1 31.00000 30 60 0.3421053
## 9 clay 1 19.00000 0 5 1.0000000
## 10 clay 1 24.79000 5 15 1.0000000
## 11 clay 1 30.20690 15 30 0.7733333
## 12 clay 1 38.38462 30 60 0.3421053# Novamente, desta vez em fatias de 1 cm, calculando quantis
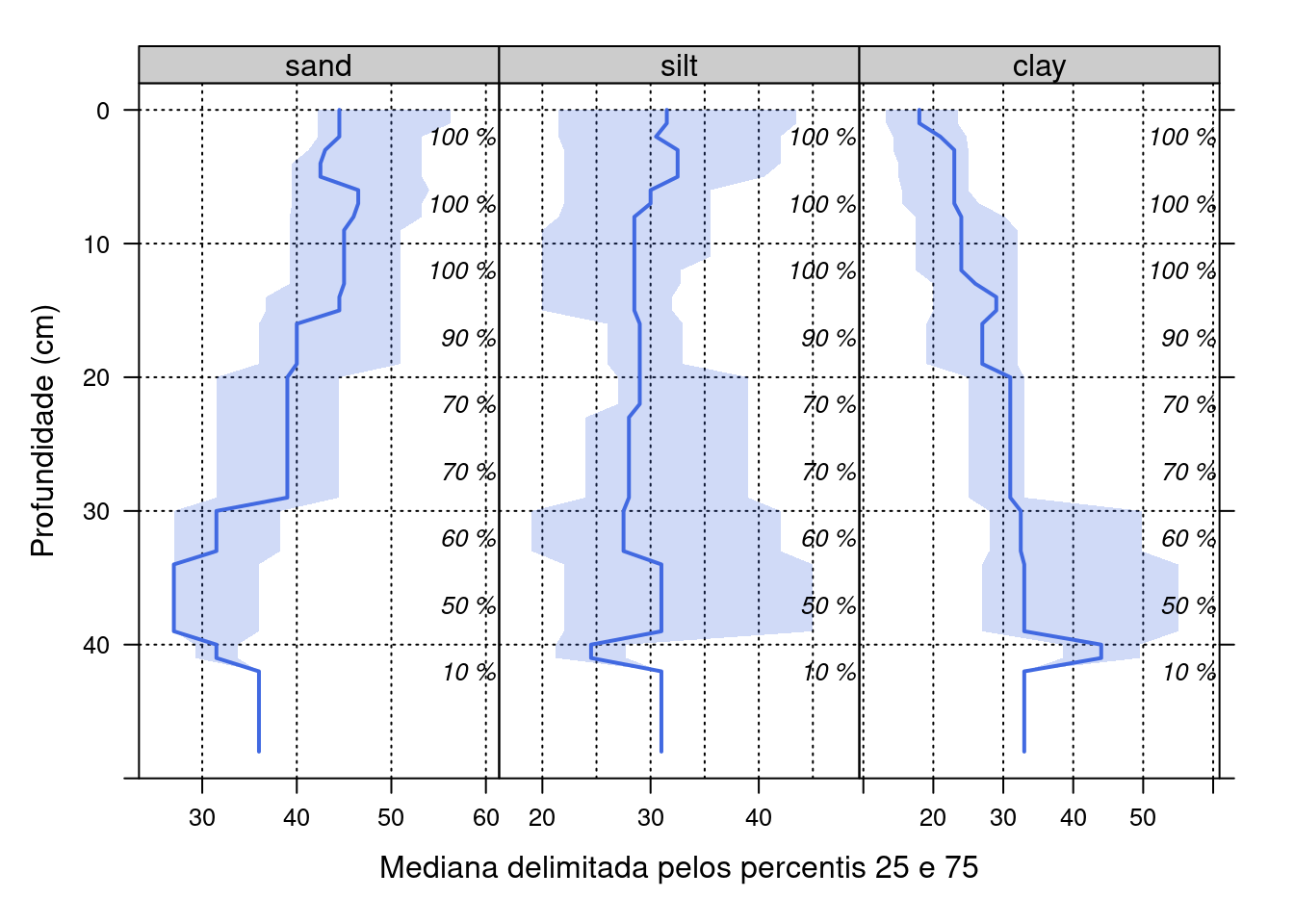
agg <- aqp::slab(sp4, fm= ~ sand + silt + clay)
# Agregando coleções de perfis de solo ao longo de períodos regulares “Slabs”
lattice::xyplot(top ~ p.q50 | variable, data=agg, ylab='Profundidade (cm)',
xlab='Mediana delimitada pelos percentis 25 e 75',
lower=agg$p.q25, upper=agg$p.q75, ylim=c(50,-2),
panel=panel.depth_function,
alpha=0.25, sync.colors=TRUE,
par.settings=list(superpose.line=list(col='RoyalBlue', lwd=2)),
prepanel=prepanel.depth_function,
cf=agg$contributing_fraction, cf.col='black', cf.interval=5,
layout=c(3,1), strip=strip.custom(bg=grey(0.8)),
scales=list(x=list(tick.number=4, alternating=3, relation='free'))
)
Análise de Dissimilaridade (Dendrogramas)
Os dendrogramas são uma maneira conveniente de descrever a dissimilaridade entre pares (pair-wise), comumente associada ao tópico da análise de cluster.
O cálculo da dissimilaridade entre perfis é realizado usando a função profile_compare(). Os valores de dissimilaridade dependem da seleção de atributos [por exemplo, argila, CEC, pH etc.], parâmetro opcional de ponderação em profundidade (k) e uma profundidade máxima de avaliação (max_d). Maiores informações dobre análise de dissimilaridade pode ser encontrado em Beaudette et al. (2013) e com exemplos de aplicação em Pinheiro et al. (2016) e Pinheiro et al. (2018).
# Dissimilaridade usando o conjunto sp4
# usando Ex-Ca:Mg e CTC a pH 7
# sem ponderação de profundidade (k = 0)
# até uma profundidade máxima de 50 cm
d <- aqp::profile_compare(sp4, vars=c('ex_Ca_to_Mg', 'CEC_7'), k=0, max_d=50)
# Verificar matriz de distância
round(d, 1)## Dissimilarities :
## colusa glenn kings mariposa mendocino napa san benito shasta
## glenn 15.8
## kings 18.0 13.1
## mariposa 17.4 20.6 25.5
## mendocino 14.1 8.2 17.0 24.5
## napa 33.5 24.9 30.5 39.3 22.2
## san benito 28.8 21.4 27.4 38.3 16.4 18.0
## shasta 19.2 13.6 8.7 26.6 17.6 34.8 23.3
## shasta-trinity 8.4 16.9 22.3 18.6 17.0 30.9 28.3 23.3
## tehama 32.1 23.9 29.2 37.6 20.8 9.0 15.3 32.7
## shasta-trinity
## glenn
## kings
## mariposa
## mendocino
## napa
## san benito
## shasta
## shasta-trinity
## tehama 29.2
##
## Metric : mixed ; Types = I, I
## Number of objects : 10# Análise hierárquica de cluster em um conjunto de dissimilaridades (Hierarchical Clustering)
h <- stats::hclust(d)
# Melhor ajuste de margens
par(mar=c(4,3,2,3))
##Plotando dendrograma
plot(as.dendrogram(h))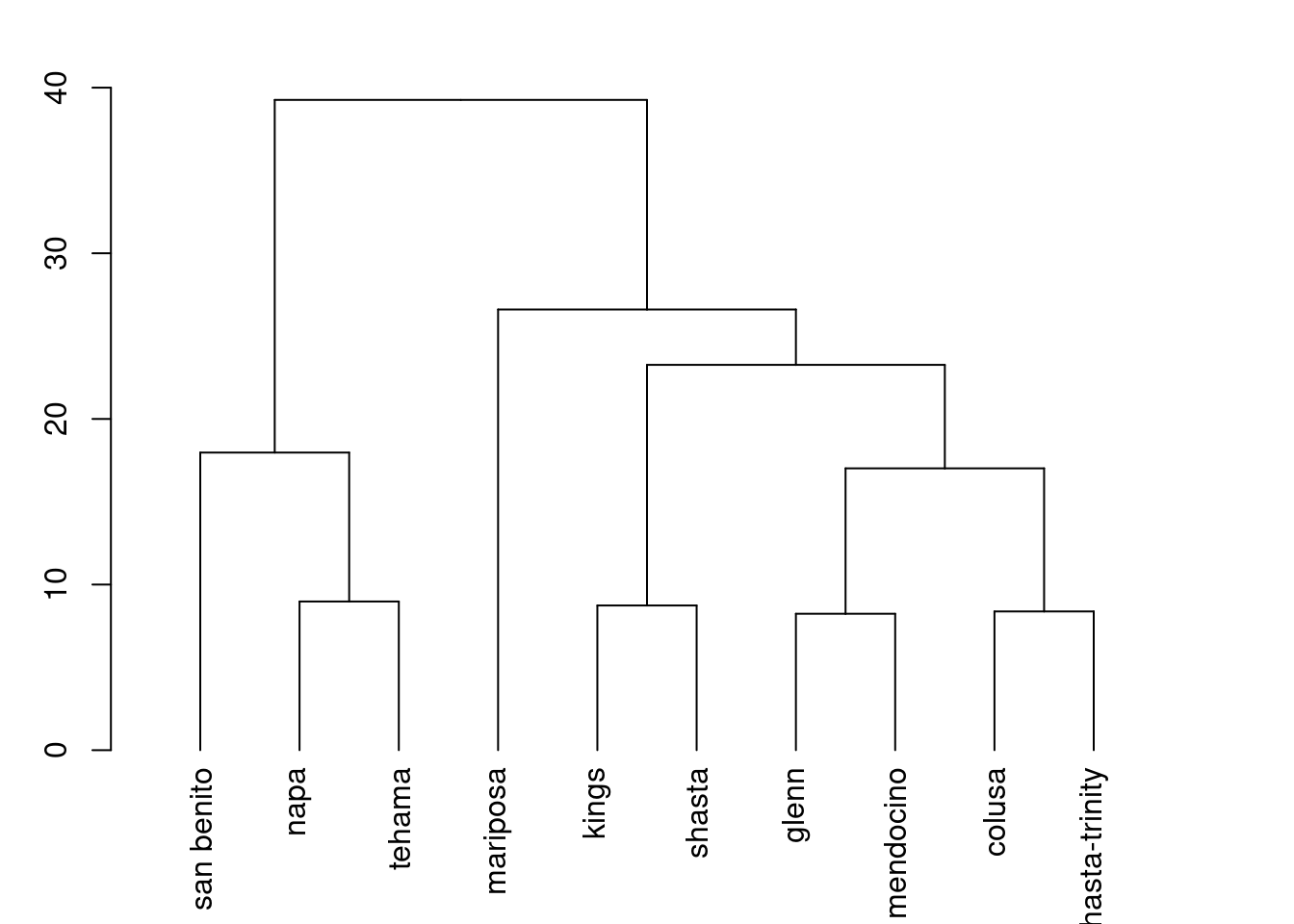
#stats::rect.hclust(h,5)
################
# Fazendo de outra forma com função diana (DIvisive ANAlysis Clustering)
# vizualize a matriz de dissimilaridade via cluster hierárquico divisivo
d.diana <- cluster::diana(d)
## Plotando Dendograma
# esta função é do pacote sharpshootR
# requer alguns ajustes manuais
par(mar=c(1,1,5,1))
sharpshootR::plotProfileDendrogram(sp4, d.diana, scaling.factor = 0.8, y.offset = 5, width=0.25, color='ex_Ca_to_Mg')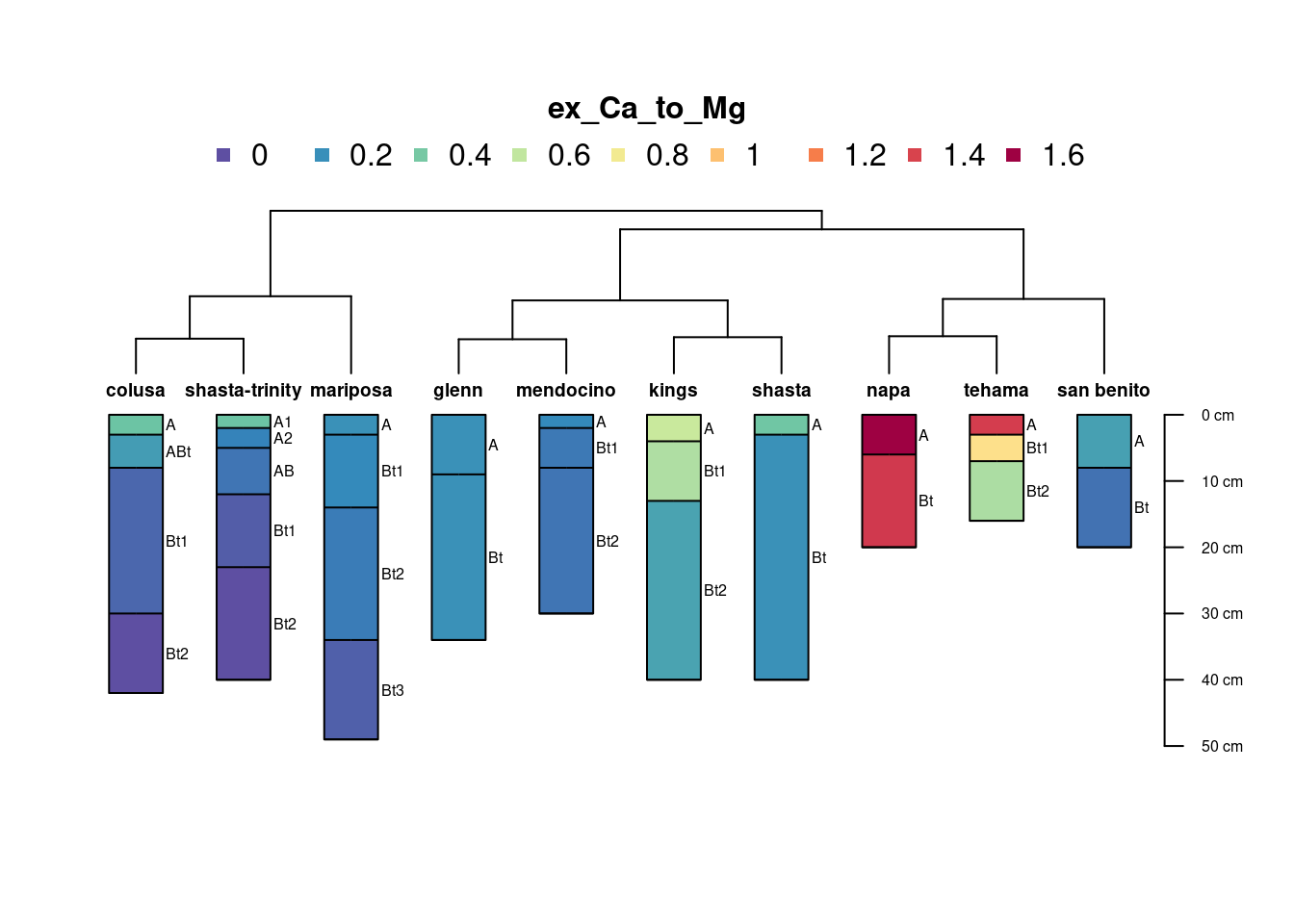
# solos de interesse do levatamento Americano
s.list <- c('hornitos', 'perkins', 'argonaut', 'inks', 'mokelumne',
'dunstone', 'auburn', 'pentz', 'pardee', 'peters',
'amador', 'laniger')
# Buscar e converter dados em um SoilProfileCollection
s <- soilDB::fetchOSD(s.list)
# Estimando a profundidade do solo com base na designação do horizontes
s$soil.depth <- profileApply(s, FUN = max)
# plot profiles, order by soil depth
par(mar=c(0, 0, 0, 0))
plot(s, name='hzname', plot.order=order(s$soil.depth), cex.names=0.85, axis.line.offset=-5, width=0.25, id.style='top')
d <- SoilTaxonomyDendrogram(s, cex.taxon.labels=0.8, width=0.25, y.offset = 0.4)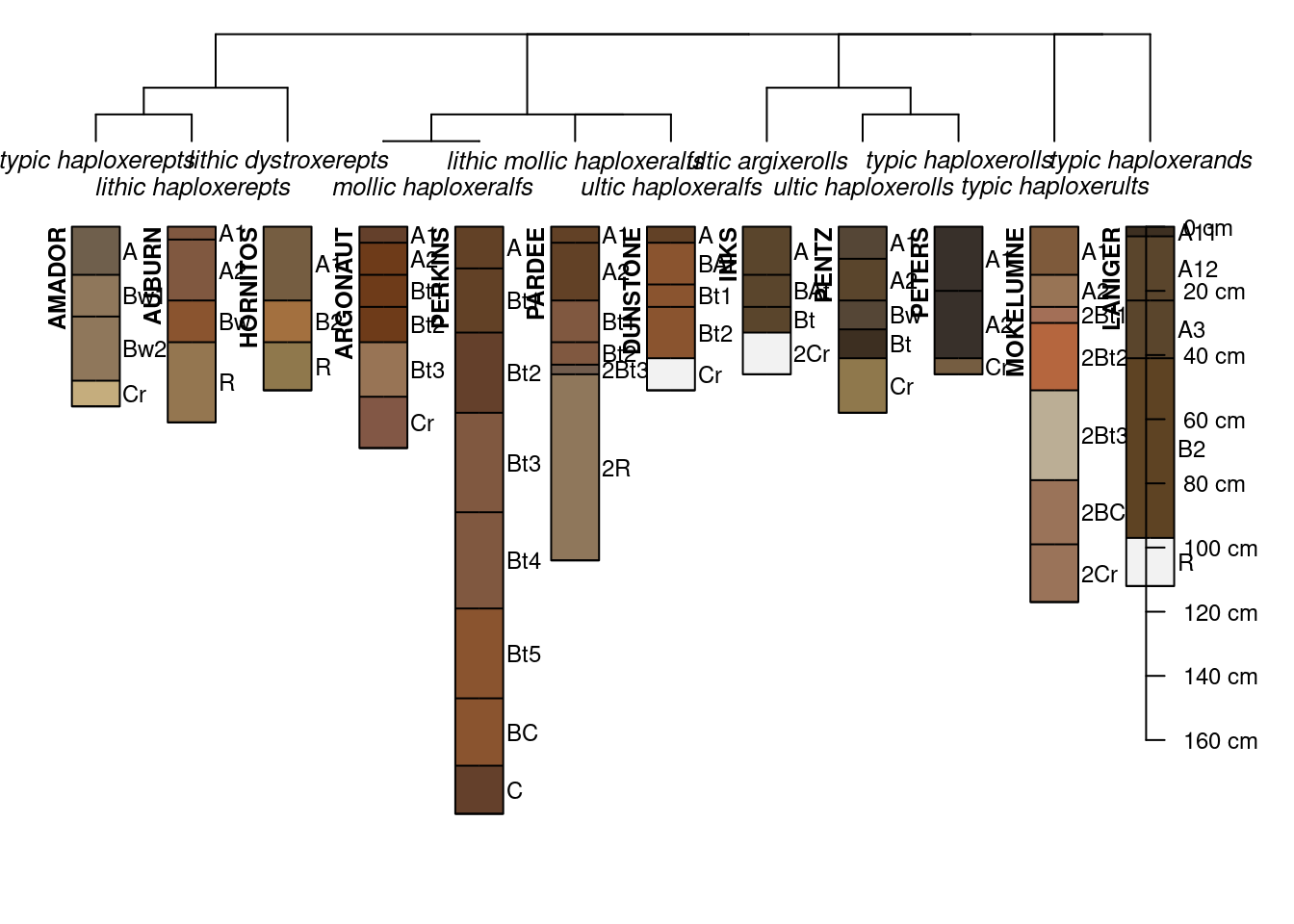
Parte III
Nessa parte é usado outra base de dados do soilDB, são eles o conjunto loafercreek e o conjunto mineralKing, e aqui além de plots que marcam os horizontes diagnósticos de alguns perfis são feitos alguns plots que relacionam o perfil e a paisagem. Maiores comentários estão na linha de código
# Carregando onconjunto de dados de solos loafercreek
data("loafercreek")
# selecionar os 10 primeiros perfis
x <- loafercreek[1:10, ]
#reconfigurando as margens porque o default é muito largo
par(mar=c(1,1,2,1))
# Alguns plots
plot(x)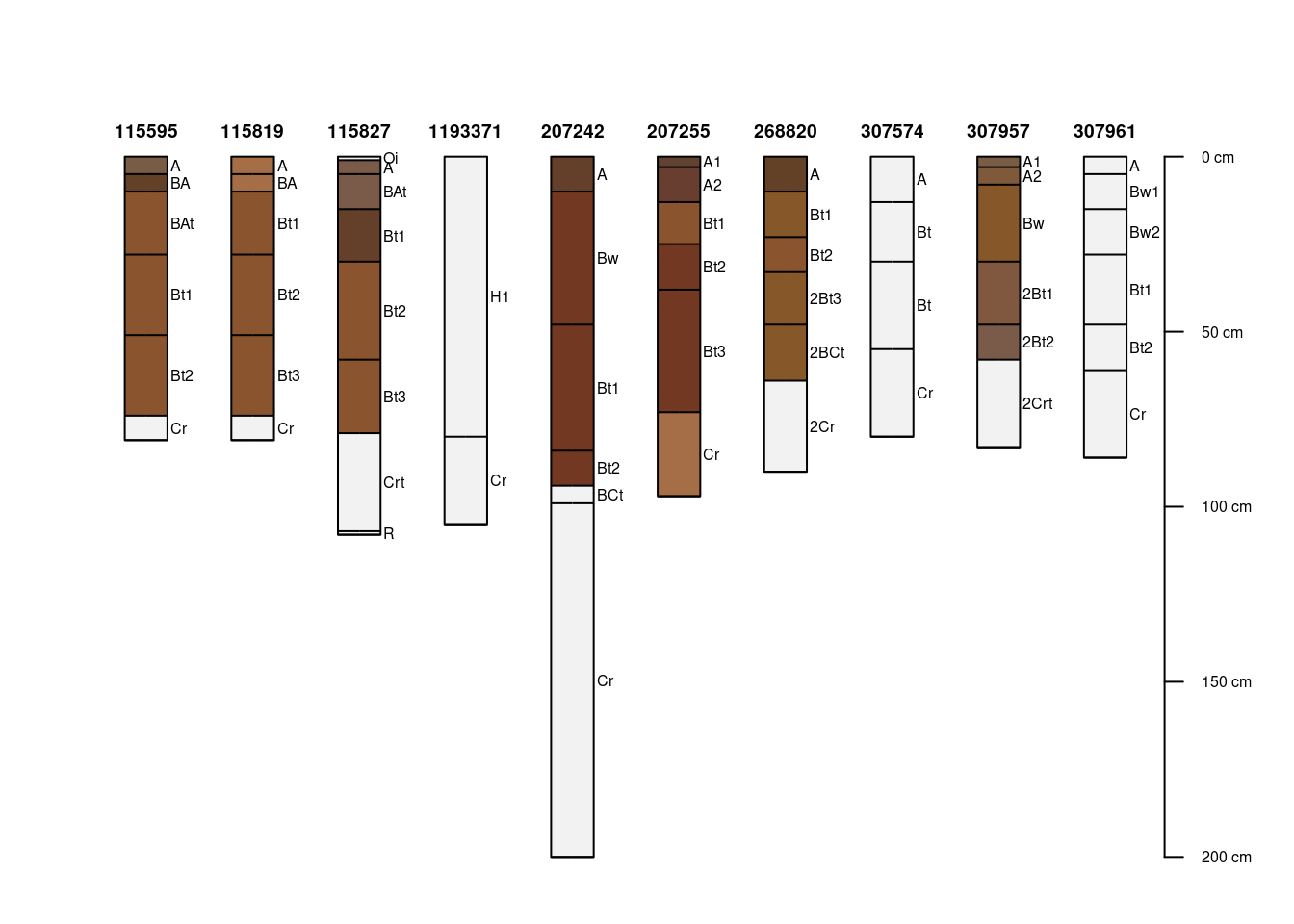
plot(x, label='pedon_id', id.style='side')
str(x)## Formal class 'SoilProfileCollection' [package "aqp"] with 9 slots
## ..@ idcol : chr "peiid"
## ..@ hzidcol : chr "phiid"
## ..@ depthcols : chr [1:2] "hzdept" "hzdepb"
## ..@ metadata :List of 8
## .. ..$ aqp_df_class : chr "data.frame"
## .. ..$ aqp_group_by : chr ""
## .. ..$ aqp_hzdesgn : chr ""
## .. ..$ aqp_hztexcl : chr ""
## .. ..$ depth_units : chr "cm"
## .. ..$ stringsAsFactors: logi FALSE
## .. ..$ original.order : int [1:56] 1 2 3 4 5 6 7 8 9 10 ...
## .. ..$ origin : chr "NASIS pedons"
## ..@ horizons :'data.frame': 56 obs. of 73 variables:
## .. ..$ phiid : chr [1:56] "579662" "579661" "579660" "579659" ...
## .. ..$ peiid : chr [1:56] "115595" "115595" "115595" "115595" ...
## .. ..$ hzname : chr [1:56] "A" "BA" "BAt" "Bt1" ...
## .. ..$ genhz : chr [1:56] NA NA NA NA ...
## .. ..$ hzdept : int [1:56] 0 5 10 28 51 74 0 5 10 28 ...
## .. ..$ hzdepb : num [1:56] 5 10 28 51 74 81 5 10 28 51 ...
## .. ..$ bounddistinct : chr [1:56] "abrupt" "clear" "clear" "clear" ...
## .. ..$ boundtopo : chr [1:56] "smooth" "wavy" "smooth" "smooth" ...
## .. ..$ clay : num [1:56] 15 20 25 29 35 NA 15 20 25 29 ...
## .. ..$ silt : num [1:56] NA NA NA NA NA NA NA NA NA NA ...
## .. ..$ sand : num [1:56] NA NA NA NA NA NA NA NA NA NA ...
## .. ..$ fragvoltot : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ texture : chr [1:56] "L" "L" "L" "CL" ...
## .. ..$ texcl : chr [1:56] "l" "l" "l" "cl" ...
## .. ..$ lieutex : chr [1:56] NA NA NA NA ...
## .. ..$ phfield : num [1:56] 5.5 4.9 5.5 6 6 ...
## .. ..$ effclass : chr [1:56] "none" "none" "none" "none" ...
## .. ..$ labsampnum : chr [1:56] "01P1566" "01P1567" "01P1568" "01P1569" ...
## .. ..$ rupresblkdry : chr [1:56] "moderately hard" "moderately hard" "slightly hard" "moderately hard" ...
## .. ..$ rupresblkmst : chr [1:56] "very friable" "very friable" "very friable" "very friable" ...
## .. ..$ rupresblkcem : chr [1:56] NA NA NA NA ...
## .. ..$ stickiness : chr [1:56] "nonsticky" "nonsticky" "slightly sticky" "slightly sticky" ...
## .. ..$ plasticity : chr [1:56] "slightly plastic" "slightly plastic" "slightly plastic" "moderately plastic" ...
## .. ..$ ksatpedon : num [1:56] NA NA NA NA NA NA NA NA NA NA ...
## .. ..$ texture_class : chr [1:56] "l" "l" "l" "cl" ...
## .. ..$ d_r : num [1:56] 0.58 0.616 0.637 0.637 0.637 ...
## .. ..$ d_g : num [1:56] 0.462 0.452 0.441 0.441 0.441 ...
## .. ..$ d_b : num [1:56] 0.313 0.218 0.248 0.248 0.248 ...
## .. ..$ d_hue : chr [1:56] "10YR" "10YR" "7.5YR" "7.5YR" ...
## .. ..$ d_value : num [1:56] 5 5 5 5 5 NA 6 6 5 5 ...
## .. ..$ d_chroma : num [1:56] 4 6 6 6 6 NA 6 6 6 6 ...
## .. ..$ d_sigma : num [1:56] NA NA NA NA NA NA NA NA NA NA ...
## .. ..$ m_r : num [1:56] 0.471 0.383 0.541 0.541 0.541 ...
## .. ..$ m_g : num [1:56] 0.359 0.257 0.331 0.331 0.331 ...
## .. ..$ m_b : num [1:56] 0.268 0.151 0.185 0.185 0.185 ...
## .. ..$ m_hue : chr [1:56] "7.5YR" "7.5YR" "5YR" "5YR" ...
## .. ..$ m_value : num [1:56] 4 3 4 4 4 NA 5 5 4 4 ...
## .. ..$ m_chroma : num [1:56] 3 4 6 6 6 NA 6 6 6 6 ...
## .. ..$ m_sigma : num [1:56] NA NA NA NA NA NA NA NA NA NA ...
## .. ..$ moist_soil_color : chr [1:56] "#785C44" "#624126" "#8A542F" "#8A542F" ...
## .. ..$ dry_soil_color : chr [1:56] "#947650" "#9D7338" "#A3703F" "#A3703F" ...
## .. ..$ fine_gravel : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ gravel : num [1:56] 0 5 5 5 0 0 0 5 5 5 ...
## .. ..$ cobbles : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ stones : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ boulders : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ channers : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ flagstones : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ parafine_gravel : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ paragravel : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ paracobbles : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ parastones : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ paraboulders : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ parachanners : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ paraflagstones : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ unspecified : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ total_frags_pct_nopf: num [1:56] 0 5 5 5 0 0 0 5 5 5 ...
## .. ..$ total_frags_pct : num [1:56] 0 5 5 5 0 0 0 5 5 5 ...
## .. ..$ art_fgr : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ art_gr : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ art_cb : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ art_st : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ art_by : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ art_ch : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ art_fl : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ art_unspecified : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ total_art_pct : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ huartvol_cohesive : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ huartvol_penetrable : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ huartvol_innocuous : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ huartvol_persistent : num [1:56] 0 0 0 0 0 0 0 0 0 0 ...
## .. ..$ soil_color : chr [1:56] "#785C44" "#624126" "#8A542F" "#8A542F" ...
## .. ..$ hzID : chr [1:56] "652" "653" "654" "655" ...
## ..@ site :'data.frame': 10 obs. of 114 variables:
## .. ..$ peiid : chr [1:10] "115595" "115819" "115827" "1193371" ...
## .. ..$ siteiid : int [1:10] 114622 159118 159056 1231724 254459 209260 269617 307647 308029 308033
## .. ..$ pedon_id : chr [1:10] "300J" "S2000CA007009" "S2000CA007012" "2017CA6306054N" ...
## .. ..$ site_id : chr [1:10] "300J" "300J" "716Q" "2017CA6306054N" ...
## .. ..$ obs_date : POSIXct[1:10], format: "1999-06-21 04:00:00" "1999-06-21 04:00:00" ...
## .. ..$ utmzone : int [1:10] 10 10 10 NA 10 10 10 10 10 10
## .. ..$ utmeasting : num [1:10] 634515 634515 633987 NA 729321 ...
## .. ..$ utmnorthing : num [1:10] 4367113 4367113 4375530 NA 4204365 ...
## .. ..$ x : num [1:10] -121 -121 -121 NA -120 ...
## .. ..$ y : num [1:10] 39.4 39.4 39.5 NA 38 ...
## .. ..$ horizdatnm : chr [1:10] "NAD83" "NAD83" "NAD83" NA ...
## .. ..$ x_std : num [1:10] -121 -121 -121 -120 -120 ...
## .. ..$ y_std : num [1:10] 39.4 39.4 39.5 37.9 38 ...
## .. ..$ gpspositionalerror : num [1:10] NA NA NA NA NA NA NA NA NA NA
## .. ..$ describer : chr [1:10] "Felix J. Panlasigui Jr." "Felix Panlasigui" "DWB" "Andrew Brown" ...
## .. ..$ pedonpurpose : chr [1:10] "full pedon description" "full pedon description" "full pedon description" NA ...
## .. ..$ pedontype : chr [1:10] "representative pedon for component" "representative pedon for component" "OSD pedon" NA ...
## .. ..$ pedlabsampnum : chr [1:10] "01N0263" "01N0263" "01N0266" NA ...
## .. ..$ labdatadescflag : int [1:10] 0 0 0 0 1 0 0 0 0 0
## .. ..$ tsectstopnum : int [1:10] NA NA NA NA NA 1 NA NA NA NA
## .. ..$ tsectinterval : num [1:10] NA NA NA NA NA 30 NA NA NA NA
## .. ..$ utransectid : logi [1:10] NA NA NA NA NA NA ...
## .. ..$ tsectkind : chr [1:10] NA NA NA NA ...
## .. ..$ tsectselmeth : chr [1:10] NA NA NA NA ...
## .. ..$ elev_field : num [1:10] 201 201 365 422 536 408 328 150 280 320
## .. ..$ slope_field : num [1:10] 8 8 18 NA 45 25 9 NA 5 3
## .. ..$ aspect_field : int [1:10] 152 152 290 NA 120 145 344 NA 90 238
## .. ..$ plantassocnm : chr [1:10] NA NA NA NA ...
## .. ..$ earthcovkind1 : chr [1:10] NA NA NA NA ...
## .. ..$ earthcovkind2 : chr [1:10] NA NA NA NA ...
## .. ..$ erocl : chr [1:10] NA NA NA NA ...
## .. ..$ bedrckdepth : int [1:10] 74 74 107 80 99 73 64 NA NA NA
## .. ..$ bedrckkind : chr [1:10] "metavolcanics" "metavolcanics" "metavolcanics" "greenstone" ...
## .. ..$ bedrckhardness : chr [1:10] NA NA NA "moderately" ...
## .. ..$ hillslopeprof : chr [1:10] NA NA NA NA ...
## .. ..$ geomslopeseg : chr [1:10] NA NA NA NA ...
## .. ..$ shapeacross : Factor w/ 5 levels "concave","linear",..: 1 1 NA NA 1 2 1 NA 2 NA
## .. ..$ shapedown : Factor w/ 5 levels "concave","linear",..: NA NA NA NA 2 2 3 NA 3 NA
## .. ..$ slopecomplex : chr [1:10] NA NA NA NA ...
## .. ..$ drainagecl : chr [1:10] NA NA "well" NA ...
## .. ..$ flodfreqcl : chr [1:10] NA NA NA NA ...
## .. ..$ floddurcl : chr [1:10] NA NA NA NA ...
## .. ..$ flodmonthbeg : chr [1:10] NA NA NA NA ...
## .. ..$ pondfreqcl : chr [1:10] NA NA NA NA ...
## .. ..$ ponddurcl : chr [1:10] NA NA NA NA ...
## .. ..$ pondmonthbeg : chr [1:10] NA NA NA NA ...
## .. ..$ geomposhill : chr [1:10] NA NA NA "side slope" ...
## .. ..$ geomposmntn : chr [1:10] NA NA NA NA ...
## .. ..$ geomposflats : chr [1:10] NA NA NA NA ...
## .. ..$ swaterdepth : int [1:10] NA NA NA NA NA NA NA NA NA NA
## .. ..$ slope_shape : Factor w/ 9 levels "concave / convex",..: NA NA NA NA 2 5 1 NA 4 NA
## .. ..$ classdate : POSIXct[1:10], format: "2012-07-24 04:02:22" "2011-07-03 09:18:06" ...
## .. ..$ classifier : chr [1:10] NA NA NA "Andrew Brown" ...
## .. ..$ classtype : chr [1:10] "correlated" "correlated" "correlated" "field" ...
## .. ..$ taxonname : chr [1:10] "LOAFERCREEK" "LOAFERCREEK" "Loafercreek" "Loafercreek" ...
## .. ..$ localphase : chr [1:10] NA NA NA NA ...
## .. ..$ taxonkind : chr [1:10] "series" "series" "series" "series" ...
## .. ..$ seriesstatus : chr [1:10] NA NA NA NA ...
## .. ..$ taxpartsize : chr [1:10] "fine-loamy" "fine-loamy" "fine-loamy" NA ...
## .. ..$ taxorder : chr [1:10] "alfisols" "alfisols" "alfisols" NA ...
## .. ..$ taxsuborder : chr [1:10] "xeralfs" "xeralfs" "xeralfs" NA ...
## .. ..$ taxgrtgroup : chr [1:10] "haploxeralfs" "haploxeralfs" "haploxeralfs" NA ...
## .. ..$ taxsubgrp : chr [1:10] "ultic haploxeralfs" "typic haploxeralfs" "ultic haploxeralfs" NA ...
## .. ..$ soiltaxedition : chr [1:10] "eighth edition" "eighth edition" "ninth edition" NA ...
## .. ..$ osdtypelocflag : int [1:10] 0 0 0 0 0 0 0 0 0 0
## .. ..$ taxmoistcl : chr [1:10] "xeric" "xeric" "xeric" NA ...
## .. ..$ taxtempregime : chr [1:10] "thermic" "thermic" "thermic" NA ...
## .. ..$ taxfamother : chr [1:10] NA NA "not used" NA ...
## .. ..$ psctopdepth : int [1:10] 10 10 15 NA 25 13 23 NA 30 28
## .. ..$ pscbotdepth : int [1:10] 61 61 65 NA 99 63 64 NA 58 61
## .. ..$ selection_method : chr [1:10] "most recent" "most recent" "most recent" "single record" ...
## .. ..$ ecositeid : chr [1:10] NA NA NA NA ...
## .. ..$ ecositenm : chr [1:10] NA NA NA NA ...
## .. ..$ ecositecorrdate : POSIXct[1:10], format: NA NA ...
## .. ..$ es_classifier : chr [1:10] NA NA NA NA ...
## .. ..$ es_selection_method : chr [1:10] NA NA NA NA ...
## .. ..$ ochric.epipedon : logi [1:10] TRUE TRUE TRUE NA TRUE TRUE ...
## .. ..$ argillic.horizon : logi [1:10] TRUE TRUE TRUE NA FALSE TRUE ...
## .. ..$ paralithic.contact : logi [1:10] TRUE TRUE TRUE NA TRUE TRUE ...
## .. ..$ lithic.contact : logi [1:10] FALSE FALSE TRUE NA FALSE FALSE ...
## .. ..$ umbric.epipedon : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ cambic.horizon : logi [1:10] FALSE FALSE FALSE NA TRUE FALSE ...
## .. ..$ mollic.epipedon : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ paralithic.materials : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ lithologic.discontinuity : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ andic.soil.properties : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ densic.contact : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ abrupt.textural.change : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ aquic.conditions : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ duripan : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ slickensides : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ redox.depletions.with.chroma.2.or.less : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ redox.concentrations : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ reduced.matrix : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ densic.materials : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ volcanic.glass : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ folistic.epipedon : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ albic.materials : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. ..$ human.transported.material : logi [1:10] FALSE FALSE FALSE NA FALSE FALSE ...
## .. .. [list output truncated]
## ..@ sp :Formal class 'SpatialPoints' [package "sp"] with 3 slots
## .. .. ..@ coords : num [1, 1] 0
## .. .. ..@ bbox : logi [1, 1] NA
## .. .. ..@ proj4string:Formal class 'CRS' [package "sp"] with 1 slot
## .. .. .. .. ..@ projargs: chr NA
## ..@ diagnostic :'data.frame': 26 obs. of 4 variables:
## .. ..$ peiid : int [1:26] 115595 115595 115595 115819 115819 115819 115827 115827 115827 115827 ...
## .. ..$ featkind: chr [1:26] "ochric epipedon" "argillic horizon" "paralithic contact" "ochric epipedon" ...
## .. ..$ featdept: int [1:26] 0 10 74 0 10 74 1 15 79 107 ...
## .. ..$ featdepb: int [1:26] 10 74 81 10 74 81 5 79 107 NA ...
## ..@ restrictions:'data.frame': 9 obs. of 8 variables:
## .. ..$ peiid : int [1:9] 115595 115819 115827 115827 207242 207255 268820 307957 307961
## .. ..$ resdept : int [1:9] 74 74 79 107 99 73 64 58 61
## .. ..$ resdepb : int [1:9] 81 81 107 NA 203 97 NA 83 86
## .. ..$ resthk_l: int [1:9] NA NA NA NA NA NA NA NA NA
## .. ..$ resthk_r: int [1:9] 7 7 NA NA 104 24 NA 25 25
## .. ..$ resthk_h: int [1:9] NA NA NA NA NA NA NA NA NA
## .. ..$ reskind : chr [1:9] "bedrock, paralithic" "bedrock, paralithic" "bedrock, paralithic" "bedrock, lithic" ...
## .. ..$ reshard : chr [1:9] NA NA "strongly" NA ...# Agrupar perfis pela posição na paisagem
#groupedProfilePlot(x, groups='hillslopeprof', label='pedon_id', id.style='side')
# Carregando onconjunto de dados de solos mineralKing
data("mineralKing")
par(mar=c(1,1,2,1))
groupedProfilePlot(mineralKing, groups='taxonname', print.id=FALSE)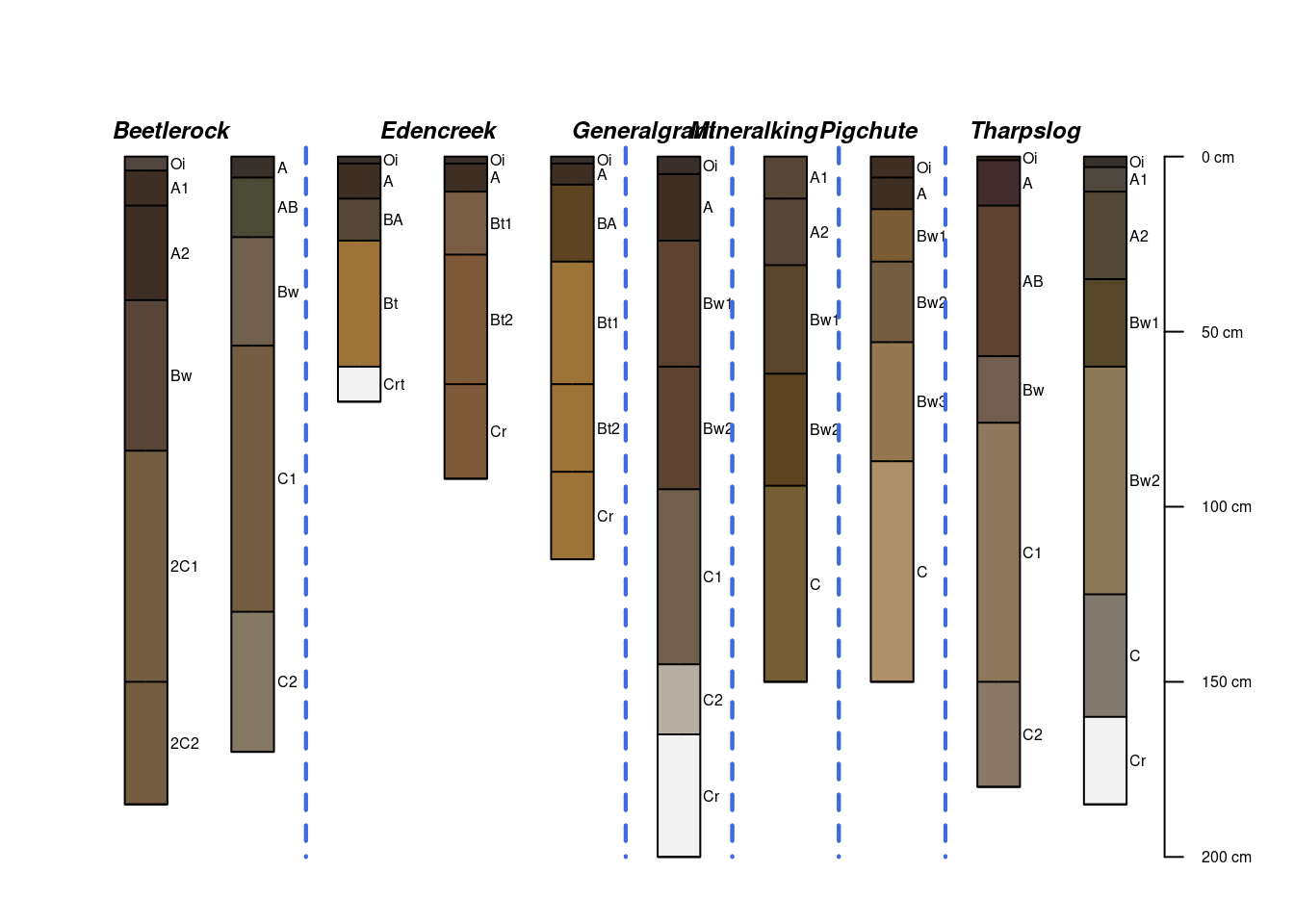
# plot de distância ao longo de um transecto
coordinates(mineralKing) <- ~ x_std + y_std
# defina o sistema de coordenada de referência (CRS)
proj4string(mineralKing) <- '+proj=longlat +datum=NAD83'
crs.utm <- CRS('+proj=utm +zone=11 +datum=NAD83') #reprojetando para utm metros
par(mar=c(4.5,4,1,1))
# plot Padrão
plotTransect(mineralKing, grad.var.name = 'elev_field', crs = crs.utm)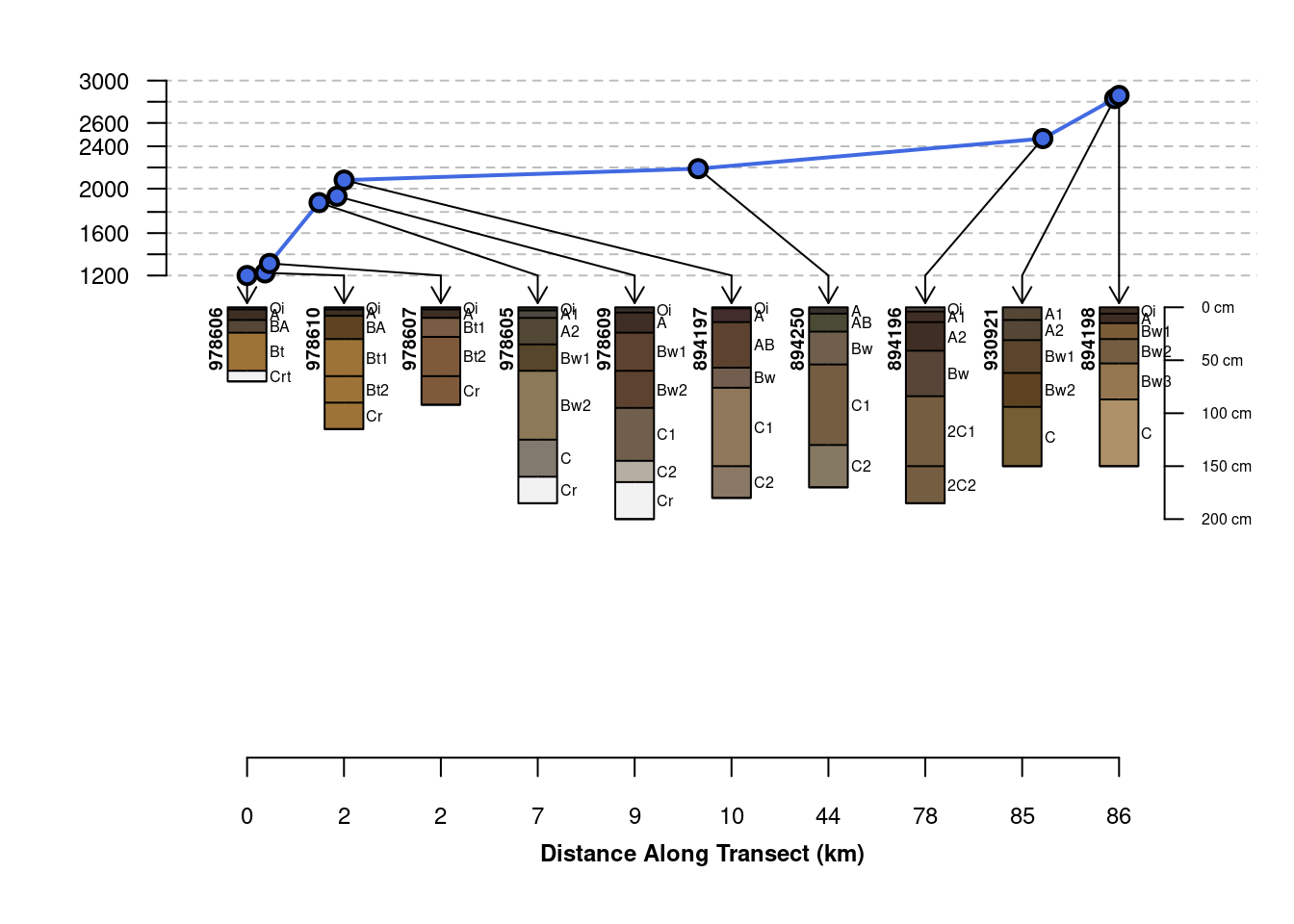
# plot com alteração de tamanho e tipo de fonte e nome dos eixos
plotTransect(mineralKing, grad.var.name='elev_field', crs=crs.utm, grad.axis.title='Elevação (m)',
distance.axis.title='Distância ao longo do transecto (km)',
label='pedon_id', scaling.factor = 0.5,
cex.names=0.8,cex.id=0.8,col.legend.cex=1.5,family="serif")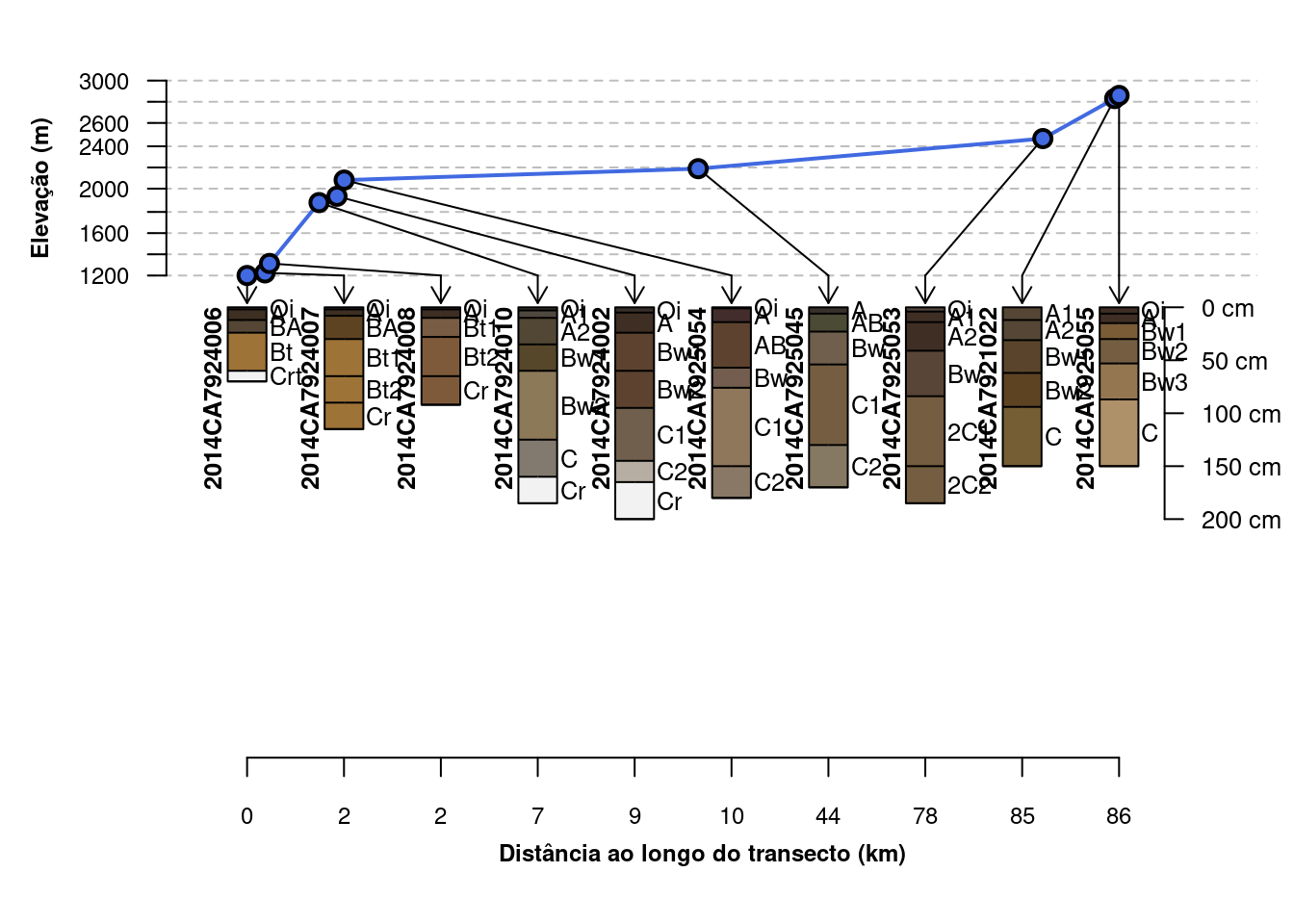 ### Se você chegou até aqui muito bem !!!! 👏👏👏
### Se você chegou até aqui muito bem !!!! 👏👏👏